Meer informatie over het gebruik van OCR-tools, Apache Spark en andere Apache Hadoop-componenten om PDF-afbeeldingen op schaal te verwerken.
Optical Character Recognition (OCR)-technologieën zijn de afgelopen 20 jaar aanzienlijk verbeterd. Gedurende die tijd is er echter weinig of geen moeite gedaan om OCR te combineren met gedistribueerde architecturen zoals Apache Hadoop om grote aantallen afbeeldingen in bijna realtime te verwerken.
In dit bericht leert u hoe u standaard open source-tools samen met Hadoop-componenten zoals Apache Spark, Apache Solr en Apache HBase kunt gebruiken om precies dat te doen voor een gebruiksgeval met informatie over medische apparatuur. U zult met name een openbare dataset gebruiken om verhalende tekst om te zetten in doorzoekbare velden.
Hoewel dit voorbeeld zich concentreert op informatie over medische apparatuur, kan het worden toegepast in veel andere scenario's waar het verwerken en bewaren van beelden vereist is. Verzekeringsmaatschappijen kunnen bijvoorbeeld al hun gescande documenten in claimbestanden doorzoekbaar maken voor een betere claimoplossing. Op dezelfde manier zou de supply chain-afdeling in een productiefaciliteit alle technische gegevensbladen van onderdelenleveranciers kunnen scannen en doorzoekbaar maken voor analisten.
Gebruiksscenario:registratie van medische hulpmiddelen
De afgelopen jaren zijn er veel veranderingen geweest op het gebied van elektronische registratie van geneesmiddelen. De ISO-standaard IDMP (Identificatie van medische producten) is zo'n berichtformaat voor het registreren van producten en de stoffen die erin zitten, waarbij de Medicinal Product ID, Packaging ID en Batch ID worden gebruikt om de producten te volgen in het geval van nadelige ervaringen, illegale import, namaak en andere kwesties van geneesmiddelenbewaking. De norm vereist dat niet alleen nieuwe producten worden geregistreerd, maar dat de oudere/gearchiveerde indiening van elk product waaraan het publiek zou kunnen worden blootgesteld, ook in elektronische vorm moet worden verstrekt.
Om te voldoen aan de IDMP-normen in verschillende bedrijven, moeten bedrijven gegevens kunnen ophalen en verwerken uit meerdere gegevensbronnen, zoals RDBMS, en in sommige gevallen ook uit oudere productgegevensbladen. Hoewel het bekend is hoe gegevens uit RDBMS kunnen worden opgenomen via technologieën zoals Apache Sqoop, vereist de verwerking van oudere documenten wat meer werk. Voor het grootste deel moeten de documenten worden opgenomen en relevante tekst moet programmatisch op grote schaal worden geëxtraheerd met behulp van bestaande OCR-technologieën.
Dataset
We zullen een dataset van de FDA gebruiken die alle 510(k)-aanvragen bevat die ooit zijn ingediend door fabrikanten van medische hulpmiddelen sinds 1976. Sectie 510(k) van de Food, Drug and Cosmetic Act vereist dat fabrikanten van apparaten die zich moeten registreren, hiervan op de hoogte stellen. FDA van hun voornemen om een medisch hulpmiddel ten minste 90 dagen van tevoren op de markt te brengen.
Deze dataset is in dit geval om verschillende redenen nuttig:
- De gegevens zijn gratis en in het publieke domein.
- De data past precies in de Europese verordening, die in juli 2016 van kracht wordt (waarbij fabrikanten moeten voldoen aan nieuwe datastandaarden). FDA-vullingen bevatten belangrijke informatie die relevant is voor het verkrijgen van een volledig beeld van IDMP.
- Het formaat van de documenten (PDF) stelt ons in staat om eenvoudige maar effectieve OCR-technieken te demonstreren bij het omgaan met documenten van meerdere formaten.
Om deze gegevens effectief te indexeren, moeten we enkele velden uit de afbeeldingen extraheren. Hieronder vindt u een voorbeelddocument met de mogelijke velden die kunnen worden geëxtraheerd.
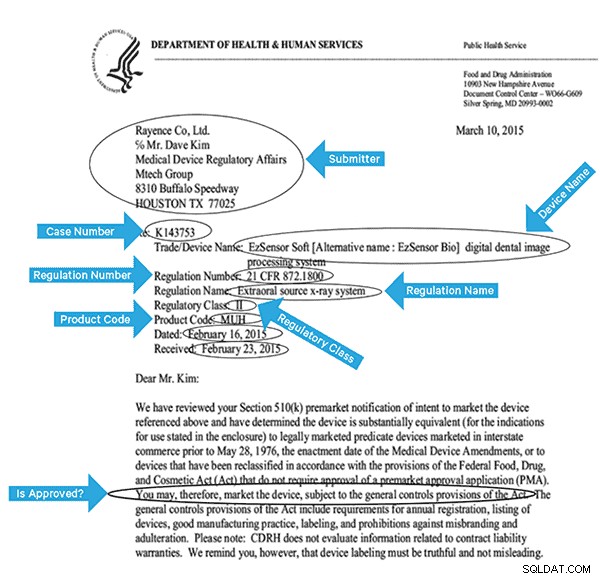
Architectuur op hoog niveau
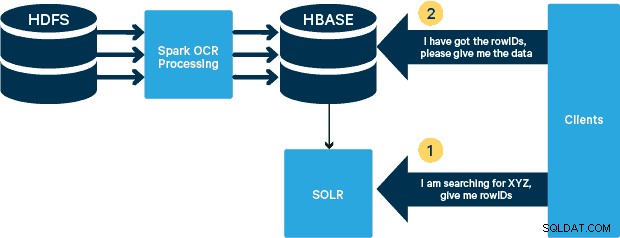
Voor dit gebruik worden de PDF's opgeslagen in HDFS en verwerkt met Spark- en OCR-bibliotheken. (De opnamestap valt buiten het bestek van dit bericht, maar het kan zo simpel zijn als het uitvoeren van hdfs -dfs -put of met behulp van een webhdfs-interface.) Spark maakt het gebruik van bijna identieke code in een Spark Streaming-toepassing mogelijk voor bijna realtime streaming, en HBase is een perfect opslagmedium voor willekeurige toegang met lage latentie - en is zeer geschikt voor het opslaan van afbeeldingen, met de nieuwe MOB-functionaliteit, om op te starten. Cloudera Search (dat bovenop Apache Solr is gebouwd) is de enige zoekoplossing die native integreert met HBase, waardoor u secundaire indexen kunt bouwen.
De tabel met medische hulpmiddelen instellen in HBase
We zullen het schema voor onze use case eenvoudig houden. De rowID is de bestandsnaam en er zijn twee kolomfamilies:"info" en "obj". De kolomfamilie "info" bevat alle velden die we uit de afbeeldingen hebben gehaald. De kolomfamilie "obj" bevat de bytes van het daadwerkelijke binaire object, in dit geval PDF. De naam van de tabel is in ons geval 'mdds'.
We zullen profiteren van de HBase MOB-functionaliteit (medium object) die is geïntroduceerd in HBASE-11339. Om HBase in te stellen om MOB te verwerken, zijn een paar extra stappen vereist, maar handig zijn instructies te vinden op deze link.
Er zijn veel manieren om de tabel programmatisch in HBase te maken (Java API, REST API of een vergelijkbare methode). Hier zullen we de HBase-shell gebruiken om de "mdds" -tabel te maken (met opzet een beschrijvende kolomfamilienaam gebruiken om dingen gemakkelijker te volgen te maken). We willen dat de kolomfamilie "info" wordt gerepliceerd naar Solr, maar niet de MOB-gegevens.
De onderstaande opdracht maakt de tabel en schakelt replicatie in op een kolomfamilie met de naam 'info'. Het is cruciaal om de optie REPLICATION_SCOPE => '1' . te specificeren , anders krijgt de HBase Lily Indexer geen updates van HBase. We willen het MOB-pad in HBase gebruiken voor objecten groter dan 10 MB. Om dat te bereiken, maken we ook een andere kolomfamilie, 'obj' genaamd, met behulp van de volgende parameters voor MOB's:
IS_MOB => true, MOB_THRESHOLD => 10240000
De IS_MOB parameter specificeert of deze kolomfamilie MOB's kan opslaan, terwijl MOB_THRESHOLD specificeert na hoe groot het object moet zijn om als een MOB te worden beschouwd. Laten we dus de tabel maken:
create 'mdds', {NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF',REPLICATION_SCOPE => '1'},{NAME => 'obj', IS_MOB => true, MOB_THRESHOLD => 10240000}
Voer de volgende opdracht uit in de HBase-shell om te bevestigen dat de tabel correct is gemaakt:
hbase(main):001:0> describe 'mdds'
Table mdds is ENABLED
mdds
COLUMN FAMILIES DESCRIPTION
{NAME => 'info', DATA_BLOCK_ENCODING => 'FAST_DIFF', BLOOMFILTER => 'ROW', REPLICATION_SCOPE => '1', VERSIONS => '1', COMPRESSION => 'NONE', MIN_VERSIONS => '0', TTL => 'FOREVER', KEEP_DELETED_CELLS => 'FALSE', BLOCKSIZE => '65536', IN_MEMORY => 'false', BLOCKCACHE => 'true'}
{NAME => 'obj', DATA_BLOCK_ENCODING => 'NONE', BLOOMFILTER => 'ROW', REPLICATION_SCOPE => '0', COMPRESSION => 'NONE', VERSIONS => '1', MIN_VERSIONS => '0', TTL => 'FOREVER', MOB_THRESHOLD => '10240000', IS_MOB => 'true', KEEP_DELETED_CELLS => 'FALSE', BLOCKSIZE => '65536', IN_MEMORY => 'false', BLOCKCACHE => 'true'}
2 row(s) in 0.3440 seconds
Gescande afbeeldingen verwerken met Tesseract
OCR heeft een lange weg afgelegd wat betreft het omgaan met lettertypevariaties, beeldruis en uitlijningsproblemen. Hier gebruiken we de open source OCR-engine Tesseract, die oorspronkelijk werd ontwikkeld als propriëtaire software in HP-labs. De ontwikkeling van Tesseract is sindsdien uitgebracht als open source software en wordt sinds 2006 gesponsord door Google.
Tesseract is een zeer draagbare softwarebibliotheek. Het gebruikt de Leptonica-beeldverwerkingsbibliotheek om een binair beeld te genereren door adaptieve drempelwaarden uit te voeren op een grijs of gekleurd beeld.
De verwerking volgt een traditionele stapsgewijze pijplijn. Hieronder volgt de ruwe stroom van stappen:
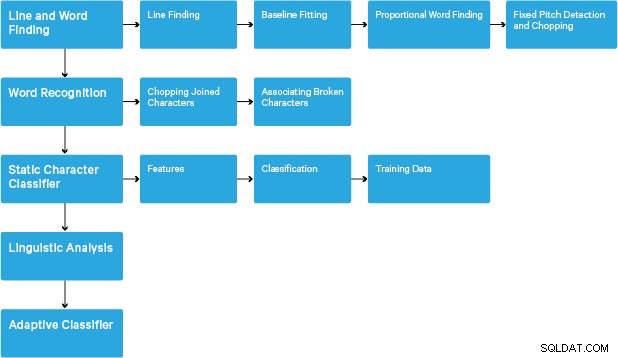
De verwerking begint met een samenhangende componentenanalyse, wat resulteert in het opslaan van de gevonden componenten. Deze stap helpt bij het inspecteren van het nesten van contouren en het aantal kind- en kleinkindcontouren.
In dit stadium worden contouren verzameld, puur door ze te nesten, in binaire grote objecten (BLOB's). BLOB's zijn georganiseerd in tekstregels en de regels en regio's worden geanalyseerd op tekst met een vaste toonhoogte of proportionele tekst. Tekstregels worden op verschillende manieren in woorden onderverdeeld, afhankelijk van het soort tekenafstand. Tekst met een vaste toonhoogte wordt onmiddellijk gehakt door karaktercellen. Proportionele tekst wordt opgedeeld in woorden met duidelijke spaties en vage spaties.
De herkenning verloopt dan als een proces van twee stappen. In de eerste doorgang wordt geprobeerd om elk woord op zijn beurt te herkennen. Elk woord dat bevredigend is, wordt als trainingsgegevens doorgegeven aan een adaptieve classificatie. De adaptieve classifier krijgt dan de kans om tekst lager op de pagina nauwkeuriger te herkennen. Omdat de adaptieve classifier mogelijk te laat iets nuttigs heeft geleerd om een bijdrage te leveren aan de bovenkant van de pagina, wordt een tweede passage over de pagina gelopen, waarin woorden die niet goed genoeg werden herkend, opnieuw worden herkend. Een laatste fase lost vage spaties op en controleert alternatieve hypothesen voor de x-hoogte om small-cap tekst te lokaliseren.
Tesseract in zijn huidige vorm is volledig unicode-compatibel en getraind voor verschillende talen. Op basis van ons onderzoek is het een van de meest nauwkeurige open source-bibliotheken die beschikbaar zijn voor OCR. Zoals eerder vermeld, gebruikt Tesseract Leptonica. We maken ook gebruik van Ghostscript om de PDF-bestanden op te splitsen in afbeeldingen. (Je kunt splitsen in een afbeeldingscompressie-indeling naar keuze; we hebben PNG gekozen.) Deze drie bibliotheken zijn geschreven in C++ en om ze vanuit Java/Scala-programma's op te roepen, moeten we implementaties van overeenkomstige Java Native Interfaces gebruiken. In ons werk gebruiken we de JNI-bindingen van JavaPresets. (De bouwinstructies zijn hieronder te vinden.) We hebben Scala gebruikt om de Spark-driver te schrijven.
val renderer :SimpleRenderer = new SimpleRenderer( ) renderer.setResolution( 300 ) val images:List[Image] = renderer.render( document )
Leptonica leest de gesplitste afbeeldingen van de vorige stap in.
ImageIO.write(
x.asInstanceOf[RenderedImage],
"png",
imageByteStream
)
val pix: PIX = pixReadMem (
ByteBuffer.wrap( imageByteStream.toByteArray( ) ).array( ),
ByteBuffer.wrap( imageByteStream.toByteArray( ) ).capacity( )
) Vervolgens gebruiken we Tesseract API-aanroepen om de tekst te extraheren. We nemen aan dat de documenten hier in het Engels zijn, vandaar dat de tweede parameter van de Init-methode "eng" is.
val api: TessBaseAPI = new TessBaseAPI( ) api.Init( null, "eng" ) api.SetImage(pix) api.GetUTF8Text().getString()
Nadat de afbeeldingen zijn verwerkt, extraheren we enkele velden uit de tekst en sturen ze naar HBase.
def populateHbase (
fileName:String,
lines: String,
pdf:org.apache.spark.input.PortableDataStream) : Unit =
{
/** Configure and open a HBase connection */
val mddsTbl = _conn.getTable( TableName.valueOf( "mdds" ));
val cf = "info"
val put = new Put( Bytes.toBytes( fileName ))
/**
* Extract Fields here using Regexes
* Create Put objects and send to HBase
*/
val aAndCP = """(?s)(?m).*\d\d\d\d\d-\d\d\d\d(.*)\nRe: (\w\d\d\d\d\d\d).*""".r
……..
lines match {
case
aAndCP( addr, casenum ) => put.add( Bytes.toBytes( cf ),
Bytes.toBytes( "submitter_info" ),
Bytes.toBytes( addr ) ).add( Bytes.toBytes( cf ),
Bytes.toBytes( "case_num" ), Bytes.toBytes( casenum ))
case _ => println( "did not match a regex" )
}
…….
lines.split("\n").foreach {
val regNumRegex = """Regulation Number:\s+(.+)""".r
val regNameRegex = """Regulation Name:\s+(.+)""".r
……..
…….
_ match {
case regNumRegex( regNum ) => put.add( Bytes.toBytes( cf ),
Bytes.toBytes( "reg_num" ),
…….
…..
case _ => print( "" )
}
}
put.add( Bytes.toBytes( cf ), Bytes.toBytes( "text" ), Bytes.toBytes( lines ))
val pdfBytes = pdf.toArray.clone
put.add(Bytes.toBytes( "obj" ), Bytes.toBytes( "pdf" ), pdfBytes )
mddsTbl.put( put )
…….
}
Als je de bovenstaande code goed bekijkt, vlak voordat we het Put-object naar HBase sturen, voegen we de onbewerkte PDF-bytes in de "obj" -kolomfamilie van de tabel in. We gebruiken HBase als opslaglaag voor zowel de geëxtraheerde velden als de onbewerkte afbeelding. Dit maakt het voor toepassingen snel en gemakkelijk om de originele afbeelding te extraheren, indien nodig. De volledige code vind je hier. (Het is vermeldenswaard dat hoewel we standaard HBase-API's gebruikten om Put-objecten voor HBase te maken, het in een echt productiesysteem verstandig zou zijn om SparkOnHBase-API's te gebruiken, die batchupdates naar HBase vanuit Spark RDD's mogelijk maken.)
Uitvoeringspijplijn
We konden elke pdf in een serieel kader verwerken. Om de verwerking te schalen, hebben we ervoor gekozen om deze pdf's gedistribueerd te verwerken met Spark. Het volgende diagram laat zien hoe we verschillende stadia van deze verwerking combineren om de workflow om te zetten in een eenvoudige macro-aanroep van Spark en om de gegevens in HBase te laden.
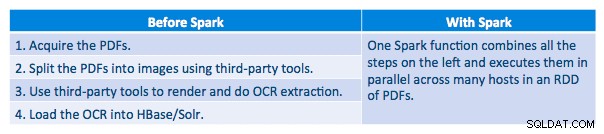
We hebben ook geprobeerd een vergelijking te maken tussen serialisatiemethoden, maar met onze dataset zagen we geen significant verschil in prestaties.
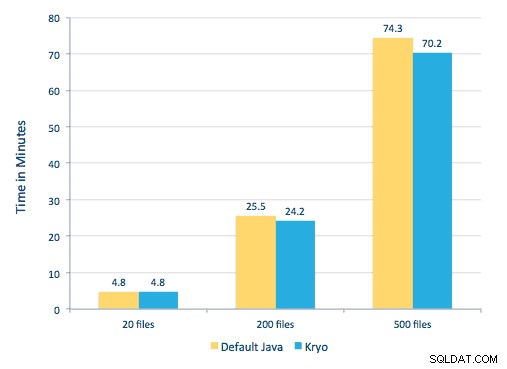
Omgeving instellen
Gebruikte hardware:cluster met vijf knooppunten met 15 GB geheugen, 4 vCPU's en 2x40 GB SSD
Omdat we C++-bibliotheken gebruikten voor verwerking, hebben we de JNI-bindingen gebruikt die hier te vinden zijn.
Bouw de JNI-bindingen voor Tesseract en Leptonica van javaCPP-presets:
-
- Op alle knooppunten:
yum -y install automake autoconf libtool zlib-devel libjpeg-devel giflib libtiff-devel libwebp libwebp-devel libicu-devel openjpeg-devel cairo-devel git clone https://github.com/bytedeco/javacpp-presets.gitcd javacpp-presets- Bouw Leptonica.
cd leptonica ./cppbuild.sh install leptonica cd cppbuild/linux-x86_64/leptonica-1.72/ LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configure make && sudo make install cd ../../../ mvn clean install cd ..
- Tesseract bouwen.
- Op alle knooppunten:
cd tesseract ./cppbuild.sh install tesseract cd tesseract/cppbuild/linux-x86_64/tesseract-3.03 LDFLAGS="-Wl,-rpath -Wl,/usr/local/lib" ./configure make && make install cd ../../../ mvn clean install cd ..
- Bouw javaCPP-voorinstellingen.
mvn clean install --projects leptonica,tesseract
We gebruiken Ghostscript om de afbeeldingen uit de PDF's te extraheren. Instructies om Ghostscript te bouwen, overeenkomend met de versies van Tesseract en Leptonica die hier worden gebruikt, zijn als volgt. (Zorg ervoor dat Ghostscript niet via de pakketbeheerder in het systeem is geïnstalleerd.)
wget https://downloads.ghostscript.com/public/ghostscript-9.16.tar.gz tar zxvf ghostscript-9.16.tar.gz cd ghostscript-9.16 ./autogen.sh && ./configure --prefix=/usr --disable-compile-inits --enable-dynamic sudo make && make soinstall && install -v -m644 base/*.h /usr/include/ghostscript && ln -v -s ghostscript /usr/include/ps (Depending on your ldpath setting, you may have to do) : sudo ln -sf /usr/lib/libgs.so /usr/local/lib/libgs.so
Zorg ervoor dat alle benodigde bibliotheken zich in het klassenpad bevinden. We plaatsen alle relevante potten in een map met de naam lib. Komma is hieronder belangrijk:
$ for i in `ls lib/*`; do export MY_JARS=./$i,$MY_JARS; done tesseract.jar, tesseract-linux-x86_64.jar, javacpp.jar, ghost4j-1.0.0.jar, leptonica.jar, leptonica-1.72-1.0.jar, leptonica-linux-x86_64.jar
We roepen het Spark programma als volgt aan. We moeten het extraLibraryPath specificeren voor native Ghostscript-bibliotheken; de andere conf is nodig voor Tesseract.
spark-submit --jars $MY_JARS --num-executors 12 --executor-memory 4G --executor- cores 1 --conf spark.executor.extraLibraryPath=/usr/local/lib --conf spark.executorEnv.TESSDATA_PREFIX=/home/vsingh/javacpp- presets/tesseract/cppbuild/1-x86_64/share/tessdata/ --conf spark.executor.extraClassPath=/etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase/ lib/htrace-core-3.1.0-incubating.jar --driver-class-path /etc/hbase/conf:/opt/cloudera/parcels/CDH/lib/hbase/lib/htrace-core-3.1.0- incubating.jar --conf spark.serializer=org.apache.spark.serializer.KryoSerializer --conf spark.kryoserializer.buffer.mb=24 --class com.cloudera.sa.OCR.IdmpExtraction
Een Solr-collectie maken
Solr integreert vrij naadloos met HBase via Lily HBase Indexer. Om te begrijpen hoe de integratie van Lily Indexer-integratie met HBase wordt gedaan, kunt u onze vorige post in het gedeelte 'HBase-replicatie en Lily HBase Indexer begrijpen' raadplegen.
Hieronder schetsen we de stappen die moeten worden uitgevoerd om de indexen te maken:
- Genereer een voorbeeldschema.xml-configuratiebestand:
solrctl --zk localhost:2181 instancedir --generate $HOME/solrcfg - Bewerk het schema.xml-bestand in
$HOME/solrcfg, met vermelding van de velden die we nodig hebben voor onze verzameling. Het volledige bestand is hier te vinden. - Upload de Solr-configuraties naar ZooKeeper:
solrctl --zk localhost:2181/solr instancedir --create mdds_collection $HOME/solrcfg - Genereer de Solr-verzameling met 2 shards (-s 2) en 2 replica's (-r 2):
solrctl --zk localhost:2181/solr --solr localhost:8983/solr collection --create mdds_collection -s 2 -r 2
In de bovenstaande opdracht hebben we een Solr-verzameling gemaakt met twee shards (-s 2) en twee replica's (-r 2) parameters. De parameters waren voldoende voor ons corpus, maar bij een daadwerkelijke implementatie zou men het aantal moeten instellen op basis van andere overwegingen die buiten ons discussiegebied hier vallen.
De indexeerder registreren
Deze stap is nodig om de Indexeer functie en HBase-replicatie toe te voegen en te configureren. Met de onderstaande opdracht wordt ZooKeeper bijgewerkt en wordt mdds_indexer toegevoegd als replicatiepeer voor HBase. Het zal ook configuraties invoegen in ZooKeeper, die Lily HBase Indexer zal gebruiken om naar de juiste collectie in Solr te verwijzen. |
hbase-indexer add-indexer -n mdds_indexer -c indexer-config.xml -cp solr.zk=localhost:2181/solr -cp solr.collection=mdds_collection.
Argumenten:
-n mdds_indexer– specificeert de naam van de indexeerder die zal worden geregistreerd in ZooKeeper-c indexer-config.xml– configuratiebestand dat het gedrag van de indexer specificeert-cp solr.zk=localhost:2181/solr– specificeert de locatie van ZooKeeper en Solr config. Dit moet worden bijgewerkt met de omgevingsspecifieke locatie van ZooKeeper.-cp solr.collection=mdds_collection– specificeert welke collectie moet worden bijgewerkt. Denk aan de Solr-configuratiestap waar we collection1 hebben gemaakt.
De index-config.xml bestand is in dit geval relatief eenvoudig; het enige dat het doet, is aan de indexeerder specificeren naar welke tabel moet worden gekeken, de klasse die als mapper zal worden gebruikt (com.ngdata.hbaseindexer.morphline.MorphlineResultToSolrMapper ), en de locatie van het Morphline-configuratiebestand. Standaard is het toewijzingstype ingesteld op rij , in welk geval het Solr-document de volledige rij wordt. Param name="morphlineFile" specificeert de locatie van het Morphlines-configuratiebestand. De locatie kan een absoluut pad zijn van uw Morphlines-bestand, maar aangezien u Cloudera Manager gebruikt, specificeert u het relatieve pad als morphlines.conf.
De inhoud van het hbase-indexer configuratiebestand is hier te vinden.
Configureren en starten van Lily HBase Indexer
Wanneer u Lily HBase Indexer inschakelt, moet u de Morphlines-transformatielogica specificeren waarmee deze Indexer updates naar de Medical Device-tabel kan parseren en alle relevante velden kan extraheren. Ga naar Services en kies Lily HBase Indexer die je eerder hebt toegevoegd. Selecteer Configuraties->Bekijken en bewerken->Service-Wide->Morphlines . Kopieer en plak het Morphlines-bestand.
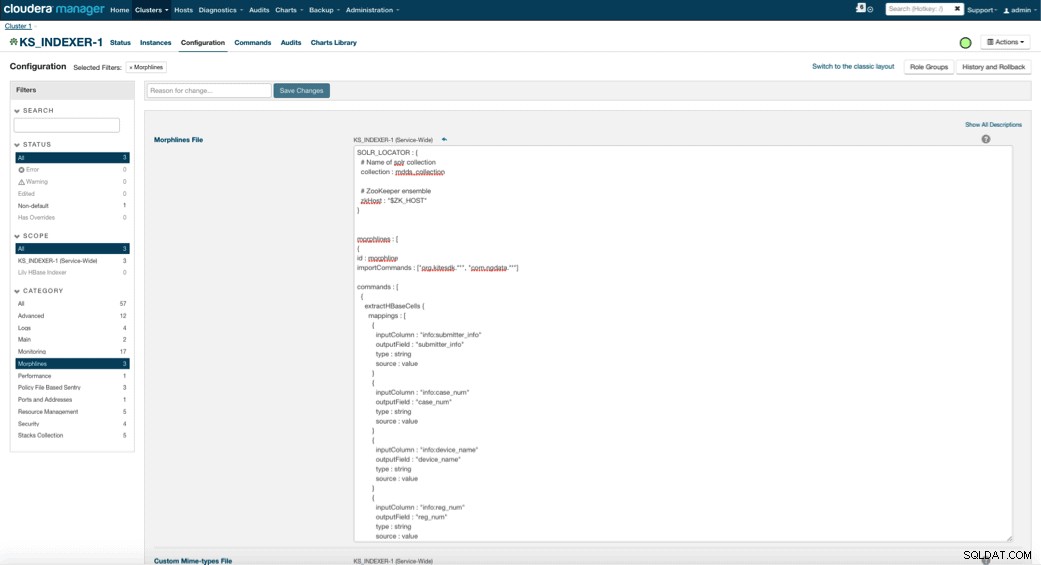
De morphlines-bibliotheek van medische apparaten zal de volgende acties uitvoeren:
- Lees de HBase-e-mailgebeurtenissen met de
extractHBaseCellscommando - Converteer de datum/tijdstempels in een veld dat Solr begrijpt, met de
convertTimestampcommando's - Laat alle extra velden weg die we niet hebben gespecificeerd in schema.xml, met de
sanitizeUknownSolrFieldscommando
Download hier een kopie van dit Morphlines-bestand.
Een belangrijke opmerking is dat het id-veld automatisch wordt gegenereerd door Lily HBase Indexer. Die instelling kan worden geconfigureerd in het bestand index-config.xml hierboven door het kenmerk unique-key-field op te geven. Het is een best practice om de standaardnaam van id te laten. Omdat deze niet is gespecificeerd in het XML-bestand hierboven, is het standaard id-veld gegenereerd en zal een combinatie zijn van RowID.
Toegang tot de gegevens
U hebt de keuze uit vele visuele hulpmiddelen om toegang te krijgen tot de geïndexeerde afbeeldingen. HUE en Solr GUI zijn beide zeer goede opties. HBase maakt ook een aantal toegangstechnieken mogelijk, niet alleen vanuit een GUI maar ook via de HBase-shell, API en zelfs eenvoudige scripttechnieken.
Integratie met Solr geeft u een grote flexibiliteit en kan ook zeer eenvoudige en geavanceerde zoekopties voor uw gegevens bieden. Als u bijvoorbeeld het Solr-schema.xml-bestand zodanig configureert dat alle velden in het e-mailobject worden opgeslagen in Solr, kunnen gebruikers via een eenvoudige zoekopdracht toegang krijgen tot volledige berichtteksten, met een afweging van opslagruimte en rekencomplexiteit. Als alternatief kunt u Solr configureren om slechts een beperkt aantal velden op te slaan, zoals de id. Met deze elementen kunnen gebruikers snel Solr doorzoeken en de rowID ophalen, die op zijn beurt kan worden gebruikt om individuele velden of de hele afbeelding uit HBase zelf op te halen.
In het bovenstaande voorbeeld wordt alleen de rowID in Solr opgeslagen, maar indexen op alle velden die uit de afbeelding zijn geëxtraheerd. Als u in dit scenario in Solr zoekt, worden HBase-rij-ID's opgehaald, die u vervolgens kunt gebruiken om HBase op te vragen. Dit type opstelling is ideaal voor Solr omdat het de opslagkosten laag houdt en volledig profiteert van de indexeringsmogelijkheden van Solr.
Voorbeeldquery's
Hieronder staan enkele voorbeeldvragen die vanuit de applicatie in Solr kunnen worden gedaan. Het idee is dat de client in eerste instantie Solr-indexen zal opvragen en de rowID van HBase retourneert. Vraag vervolgens naar HBase voor de rest van de velden en/of de originele onbewerkte afbeelding.
- Geef me alle documenten die zijn ingediend tussen de volgende data:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=received:[2010-01-06T23:59:59.999Z TO 2010-02-06T23:59:59.999Z]
- Geef me documenten die zijn ingediend onder de naam van de regelgevende instantie voor mobiele röntgensystemen:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=reg_name:Mobile x-ray system
- Geef me alle documenten die zijn ingediend door Chinese fabrikanten:
https://hbase-solr2-1.vpc.cloudera.com:8983/solr/mdds_collection/select?q=submitter_info:*China*
De id's uit Solr-documenten zijn de rij-id's in HBase; het tweede deel van de vraag is naar HBase om de gegevens te extraheren (inclusief de onbewerkte PDF indien nodig).
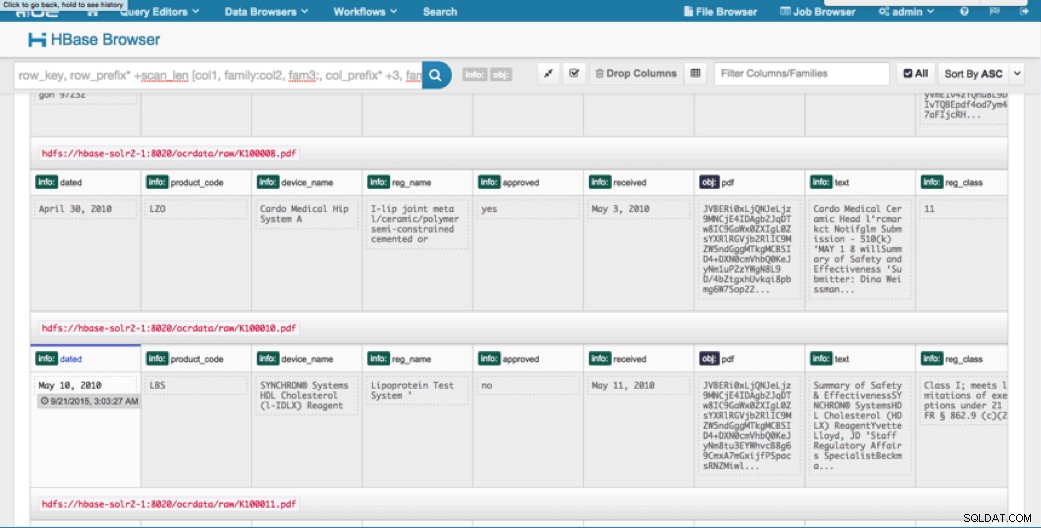
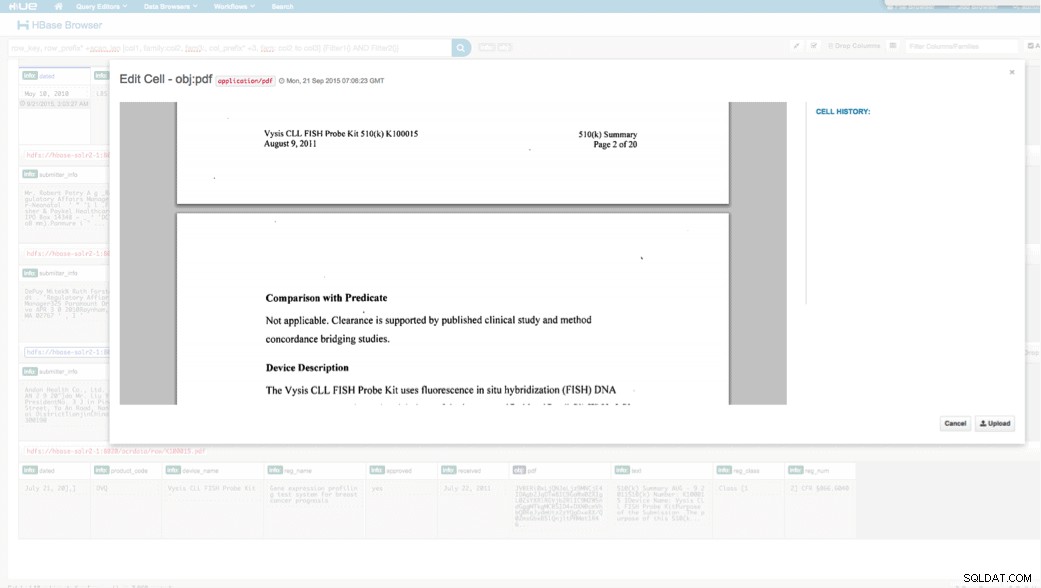
Toegang via HUE
De geüploade gegevens kunnen we bekijken via de HBase Browser in HUE. Een groot voordeel van HUE is dat het de binaire bestanden voor PDF kan detecteren en weergeven wanneer erop wordt geklikt.
Hieronder ziet u een momentopname van de weergave van de geparseerde velden in HBase-rijen en ook een weergegeven weergave van een van de PDF-objecten die zijn opgeslagen als een MOB in de obj-kolomfamilie.
Conclusie
In dit bericht hebben we laten zien hoe we standaard open source-technologieën kunnen gebruiken om OCR uit te voeren op gescande documenten met behulp van een schaalbaar Spark-programma, opslaan in HBase voor snel ophalen en indexeren van de geëxtraheerde informatie in Solr. Het moet duidelijk zijn dat:
- Gezien het berichtspecificatie-formaat kunnen we velden en waardeparen extraheren en doorzoekbaar maken via Solr.
- Deze velden uit gegevens kunnen voldoen aan de IDMP-vereisten om de oude gegevens elektronisch te maken, die ergens volgend jaar van kracht worden.
- De velden en onbewerkte afbeeldingen kunnen worden bewaard in HBase en zijn toegankelijk via standaard API's.
Als u gescande documenten moet verwerken en de gegevens moet combineren met verschillende andere bronnen in uw onderneming, overweeg dan een combinatie van Spark, HBase, Solr, Tesseract en Leptonica. Het kan u een aanzienlijke hoeveelheid tijd en geld besparen!
Jeff Shmain is Senior Solution Architect bij Cloudera. Hij heeft meer dan 16 jaar ervaring in de financiële sector met een goed begrip van effectenhandel, risico's en regelgeving. De afgelopen jaren heeft hij gewerkt aan verschillende use case-implementaties bij 8 van de 10 grootste investeringsbanken ter wereld.
Vartika Singh is Senior Solution Consultant bij Cloudera. Ze heeft meer dan 12 jaar ervaring in toegepaste machine learning en softwareontwikkeling.
